DeepMind, protein katlayıcı yapay zekasını açık kaynak yaptı
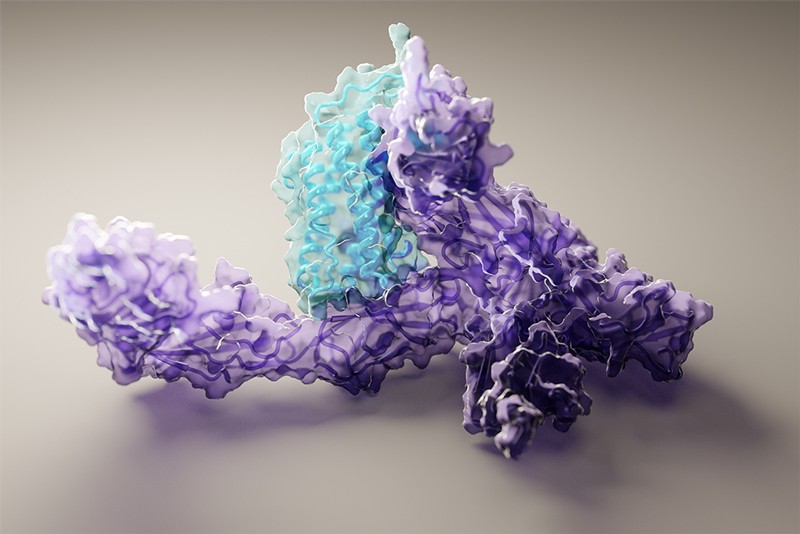 Makine öğrenimi yazılımı tarafından tahmin edildiği gibi, reseptörüne bağlı insan interlökin-12 proteininin yapısı. Kredi: Ian Haydon, UW Medicine Institute for Protein Design
Makine öğrenimi yazılımı tarafından tahmin edildiği gibi, reseptörüne bağlı insan interlökin-12 proteininin yapısı. Kredi: Ian Haydon, UW Medicine Institute for Protein Design
İnsanlar için protein yapısı tahminidir. Proteinlerin 3D şeklini doğru bir şekilde belirleyen yazılım, bilim insanlarının kullanımına geniş çapta sunulacak.
15 Temmuz’da Londra merkezli DeepMind şirketi, derin öğrenme sinir ağı AlphaFold 2’nin açık kaynaklı bir versiyonunu yayınladı ve yaklaşımını Nature’deki bir makalede açıkladı.
Bu arada, bir akademik ekip, bilim insanları arasında zaten popülerlik kazanan AlphaFold 2’den esinlenerek kendi protein tahmin aracını geliştirdi. RoseTTaFold adı verilen bu sistem, neredeyse AlphaFold 2 kadar iyi performans gösteriyor.
Illinois’deki Chicago Üniversitesi’nde hesaplamalı biyolog olan Jinbo Xu, araçları açık kaynaklı yapmanın, bilim camiasına daha da güçlü ve kullanışlı yazılımlar oluşturmak için yardım ettiği anlamına geldiğini söylüyor.
İşlevsel yapılar
Proteinler, 3 boyutlu şekillere katlandığında bu proteinlerin hücrelerdeki işlevini belirleyen amino asit dizilerinden yapılır. Onlarca yıldır araştırmacılar, protein yapılarını belirlemek için X-ışını kristalografisi ve kriyo-elektron mikroskobu gibi deneysel teknikleri kullandılar. Ancak bu tür yöntemler zaman alıcı ve maliyetli olabilir ve bazı proteinler bu tür analizlere uygun değildir.
DeepMind, yazılımının yalnızca proteinlerin sırasını (DNA tarafından belirlenir) kullanarak birçok proteinin yapısını doğru bir şekilde tahmin edebildiğini gösterdiğinde, geçen yıl bilim dünyasına şok dalgaları gönderdi. Araştırmacılar on yıllardır bu zorluk üzerinde çalışıyorlardı ve AlphaFold 2, CASP adlı iki yılda bir yapılan bir protein tahmin alıştırmasında o kadar iyi performans gösterdi ki, yarışmanın kurucu ortağı “bir anlamda sorunun çözüldüğünü” ilan etti.
Çalışmaları konusunda ketum olmakla ün yapmış DeepMind, 1 Aralık’ta CASP’de yaptığı kısa bir sunumda AlphaFold 2’yi anlattı. Ağı daha ayrıntılı olarak özetleyen bir makale yayınlamaya ve yazılımı araştırmacıların kullanımına sunmaya söz verdi, ancak başka bir şey söylemedi.
Ekibi RoseTTaFold’u geliştiren Seattle’daki Washington Üniversitesi’nde biyokimyacı olan David Baker, “Akademisyenler arasında oldukça fazla kıyamet ve kasvet vardı” diyor. “Birisi üzerinde çalıştığınız sorunu çözdüyse ancak nasıl yaptığını açıklamıyorsa, üzerinde çalışmaya nasıl devam edeceksiniz?”
Baker’ın ekibinin bir üyesi olan hesaplamalı kimyager Minkyung Baek, “O zamanlar işimi kaybetmiş gibi hissettim” diyor. Ancak DeepMind’in sunumu, Baek’in keşfetmek için sabırsızlandığı yeni fikirleri de teşvik etti. Böylece o, Baker ve meslektaşları AlphaFold 2’nin başarısını tekrarlamak için beyin fırtınası yapmaya başladılar.
Ağın, araştırmacıların tahmin etmeye çalıştığı hedeflerle evrimsel olarak ilişkili proteinler hakkındaki bilgileri nasıl kullandığı ve bir proteinin bir parçasının tahmin edilen yapılarının, ağın diğer bölümlerine karşılık gelen dizileri nasıl ele aldığını nasıl etkileyebileceği de dahil olmak üzere birkaç önemli ilerleme belirlediler.
RoseTTaFold yalnızca AlphaFold 2 kadar iyi bir performans sergilemekle kalmadı, aynı zamanda diğer CASP girişlerinden (Baker laboratuvarından bazıları dahil) çok daha iyi performans gösterdi. Baek, AlphaFold 2’ye neden eşit olamayacağı henüz belli değil, ancak bir olasılık DeepMind’in uzmanlığı, diyor Baek. “Laboratuvarımızda derin öğrenen mühendislerimiz yok.” Xu, Baek, Baker ve işbirlikçilerinin çabalarından etkilendi ve DeepMind’in başarısının mühendislik uzmanlığına ve üstün bilgi işlem gücüne erişiminden kaynaklandığından şüpheleniyor.
Hızlı yapılar
DeepMind ayrıca AlphaFold 2’yi de sorunsuz hale getirdi. AlphaFold’un baş araştırmacısı John Jumper, ağın bazı CASP girişleri için yapılar oluşturmak için günlerce bilgi işlem süresi almasına karşın, açık kaynaklı sürümün yaklaşık 16 kat daha hızlı olduğunu söylüyor. Proteinin boyutuna bağlı olarak dakikalar veya saatler içinde yapılar oluşturabilir. Bu, RoseTTaFold’un hızıyla karşılaştırılabilir.
AlphaFold 2’nin kaynak kodu ticari kuruluşlar da dahil olmak üzere ücretsiz olarak mevcut olsa da, teknik uzmanlığı olmayan araştırmacılar için henüz özellikle yararlı olmayabilir. DeepMind’in bilim için AI başkanı Pushmeet Kohli, DeepMind’in merkezi Cenevre, İsviçre’de bulunan ve kar amacı gütmeyen Drugs for Neglected Diseases girişimi de dahil olmak üzere belirli araştırmacılar ve kuruluşlarla belirli hedefleri tahmin etmek için işbirliği yaptığını, ancak erişimi genişletmeyi umduğunu söylüyor. “Bu alanda yapmak istediğimiz daha çok şey var.”
Baker’ın ekibi, RoseTTaFold kodunu ücretsiz olarak kullanılabilir hale getirmenin yanı sıra, araştırmacıların bir protein dizisini bağlayabileceği ve tahmin edilen bir yapı elde edebileceği bir sunucu kurdu. Baker, sunucunun geçen ay piyasaya sürülmesinden bu yana yaklaşık 500 kişi tarafından sunulan 5.000’den fazla proteinin yapısını tahmin ettiğini söylüyor.
Artık hem RoseTTaFold hem de AlphaFold 2 için ücretsiz olarak kullanılabilen kodla, araştırmacılar her iki gelişmeyi de geliştirebilecekler, diyor Xu ve belki de teknikleri AlphaFold 2’nin şimdiye kadar tahmin etmekte zorlandığı protein yapılarına uygun hale getirebilir. İki yoğun ilgi alanı, etkileşime giren çoklu proteinlerin komplekslerinin yapısını tahmin etmek ve yazılımı yeni proteinlerin tasarımına uygulamaktır.





Yorum yapın